Cell子刊:魏文胜团队利用碱基编辑器筛选绘制DNA损伤应答功能元件图谱
来源:生物世界 2024-12-18 14:33
该研究通过碱基编辑器筛选,绘制了DNA损伤应答功能元件图谱。
北京大学、北大-清华生命科学联合中心魏文胜团队在 Cell Reports 期刊发表了题为:Mapping functional elements of the DNA damage response through base editor screens 的研究论文。该研究通过碱基编辑器筛选,绘制了DNA损伤应答功能元件图谱。

基于团队此前开发的赖氨酸功能位点筛选策略和基因敲除策略,他们分别利用腺嘌呤碱基编辑器(ABE)和胞嘧啶碱基编辑器(CBE),对全基因组范围内的赖氨酸位点或基因进行靶向编辑,实现赖氨酸位点突变或敲除基因。随后在人类视网膜色素上皮细胞系(RPE1)中进行筛选,并结合两种DNA损伤药物处理,最终筛选出多个在DNA损伤修复过程中具有关键作用的氨基酸位点及基。
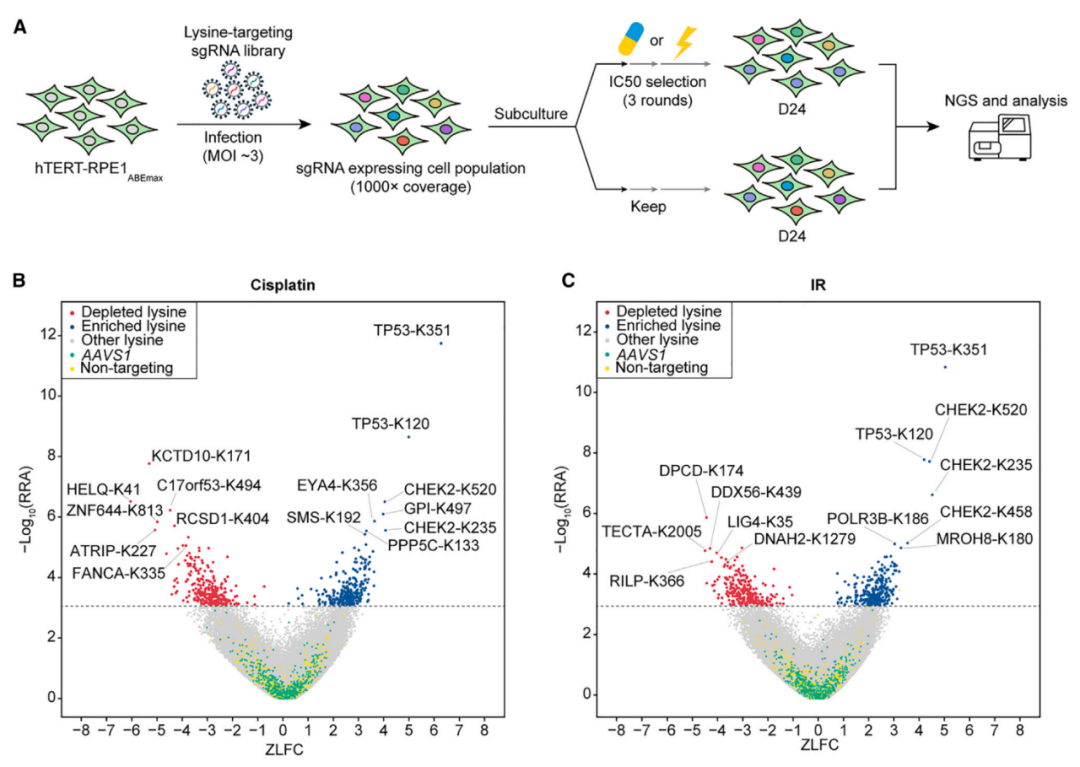
图1. 筛选流程和结果
在Cisplatin筛选结果中,负向排名前十的基因大多为已知FA修复通路的关键因子(如FANCM、FANCA、FANCG等)。研究发现其中STK35基因从未被发现与DNA损伤应答相关。文献显示,其家族成员STK19和STK11分别参与TC-NER途径和UVB诱导损伤修复。后续实验证实,STK35是一个潜在的DNA修复因子,在DNA修复过程中发挥重要作用。
2019年,C17orf53被鉴定为DNA链间交联(ICL)修复的关键因子。在该研究中,筛选结果表明C17orf53的K494位点是其在DNA损伤修复中的关键氨基酸位点。K494的突变破坏了C17orf53与其上游因子RPA的相互作用,导致严重的ICL修复缺陷、G2/M细胞周期停滞以及对顺铂治疗的敏感性显著增强。
此外,研究还鉴定出多个与DNA损伤修复相关的重要翻译后修饰位点,例如p53的K120是调控其凋亡功能的重要乙酰化位点,该位点突变可导致细胞对DNA损伤药物的高度耐受。
总结来说,该研究通过多种碱基编辑筛选策略,系统性地绘制了影响DNA损伤应答的功能性赖氨酸位点及相关基因图谱,为DNA损伤应答基因功能研究提供了全新视角,也有助于加速癌症治疗中的耐药性变异研究(图2)。
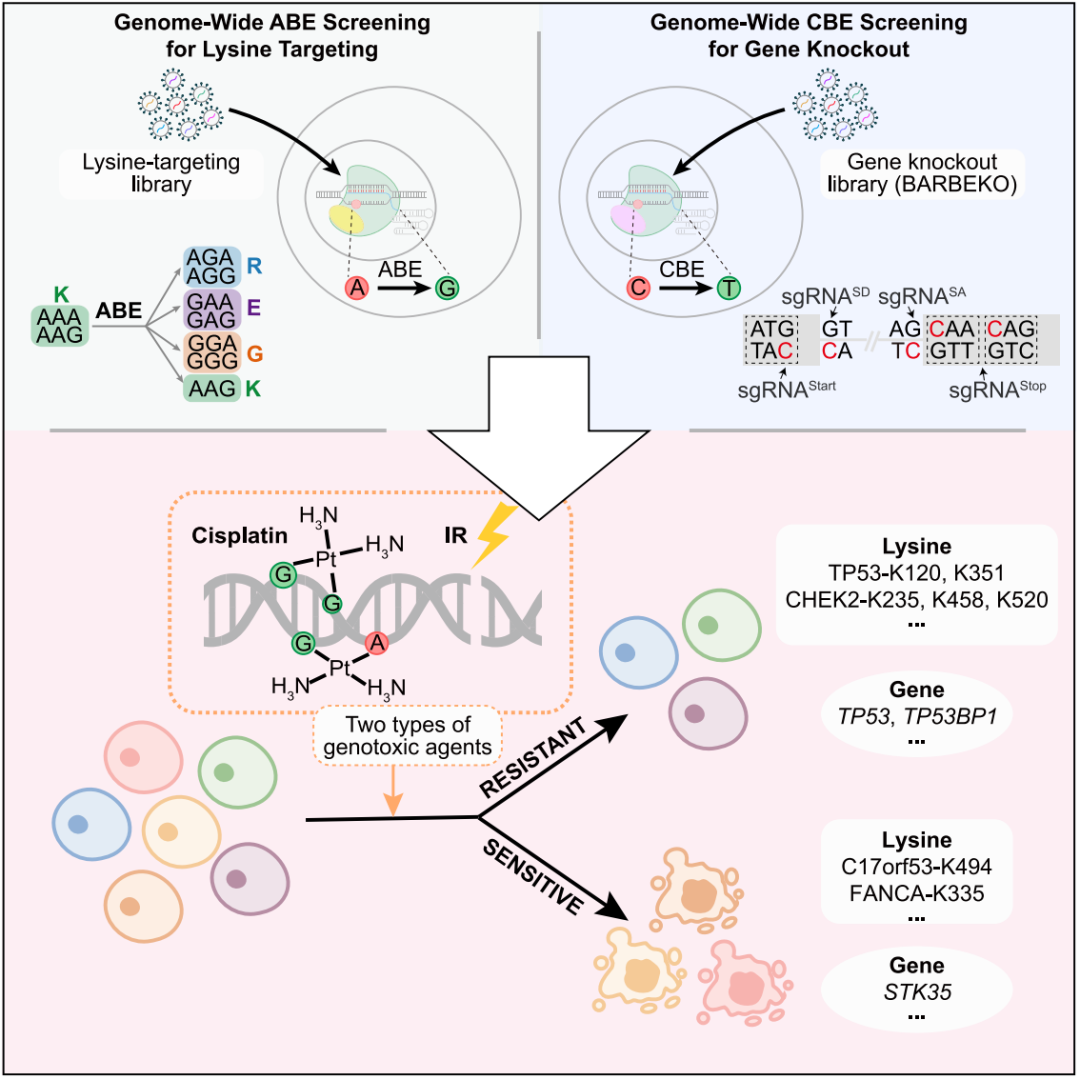
图2. 总结图
北京大学、北大-清华生命科学联合中心魏文胜课题组潘倩博士(已毕业)与博士研究生张芷瑄为论文共同第一作者。博士研究生熊杨芳、博士后宝颖、博士研究生陈天欣和许萍博士(已毕业)等人亦作出了重大贡献。该研究获得了国家自然科学基金、北大-清华生命科学联合中心及昌平实验室的资助。
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


